浅谈CRISPR/Cas12基因编辑技术
CRISPR/Cas12系统于2018年被Jennifer Doudna团队挖掘鉴定并在发表于Science杂志。CRISPR/Cas12系统由单一的RuvC结构域在远PAM端介导DNA切割,标签基因为Cas12,有的由单个crRNA介导,如CRISPR/Cas12a;有的由crRNA和tracrRNA共同介导(或融合后的sgRNA单独介导),如CRISPR/Cas12b[1,2]。
CRISPR/Cas12系统
CRISPR/Cas12系统的标签基因已有多达11种亚型(Cas12a-k)被鉴定,最近发现的CasX归类为Cas12e、CasY归类为Cas12d、Cas14归类为Cas12f、Casφ归类为Cas12j;其中研究最广泛的是Cas12a、Cas12b以及Cas12f[1]。
●Cas12a
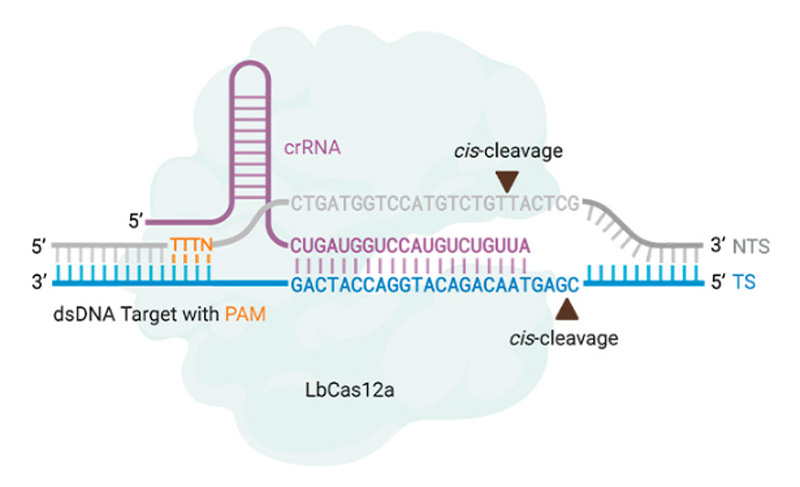
Cas12a(又称Cpf1)是一类由单个crRNA介导的核酸内切酶,能特异性识别并剪切带PAM(5-TTTN-3'或5'-TTN-3)的双链DNA(dsDNA)靶标,使DNA双链断裂并生成黏性末端。对单链DNA(ssDNA)靶标的识别和剪切不依赖PAM序列。目前最常用的Cas12a来自氨基酸球菌属的BV3L6菌株(AsCas12a)和毛螺科细菌(LbCas12a)[3]。
●Cas12b
Cas12b(又称C2c1)是一类由crRNA和tracrRNA共同介导(或融合体sgRNA单独介导)的核酸内切酶,最常用的Cas12b来源于嗜酸耐热菌 Alicyclobacillus acidoterrestris (AacCas12b)。Cas12b比Cas12a更加耐热,能特异性识别并剪切带PAM(5'-TTN-3)的dsDNA,使DNA双链断裂并生成黏性末端。同样,Cas12b特异性的剪切ssDNA靶标不依赖PAM序列。此外,双链或单链DNA靶标均能激活AacCas12b的反式剪切活性(即旁路剪切活性/附属剪切活性),即当AacCas12b酶与sgRNA、靶标DNA结合形成三元复合物后,便会被激活针对非特异序列ssDNA的反式剪切活性,可无差别地剪切反应体系中任意序列的ssDNA。

●Cas12f
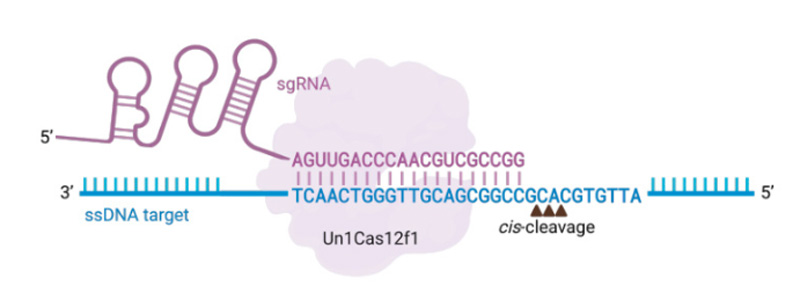
Cas12f(又名Cas14a1)是依赖于crRNA和tracrRNA共同介导的(或融合后的sgRNA单独介导的)DNA核酸内切酶,能以PAM依赖的方式特异地剪切靶标dsDNA,使DNA双链断裂并生成黏性末端。与其他Cas12蛋白类似,Cas12f蛋白同样拥有针对ssDNA的反式剪切活性,不受PAM位点的限制,且双链或单链DNA靶标均能激活Cas12f的反式剪切活性。此外,相比于其他的Cas12蛋白,Cas12f蛋白的分子量普遍较小(400-700AA)。
◆CRISPR/Cas12基因编辑原理(以CRISPR/Cas12a为例)
●CRISPR/Cas12a的顺式剪切
由单个crRNA引导,Cas12a催化自身的前体,生成成熟的crRNA,当crRNA-Cas12a复合物形成时,识别DNA非靶标链中特定的PAM位点,Ruvc结构域在距离PAM位点8nt处切割DNA靶标链,产生5’黏性末端[4]。其中LbCas12a和AsCas12a均偏好识别5’-TTTN-3’的PAM序列,弗兰西斯菌来源的Cas12a(FnCas12a)识别的PAM序列可扩展为5’-KYTV-3’(K指代T、G;Y指代C、T;V指代A、C、G)。Gao等基于结构导向的靶向诱变技术,设计出了两种AsCas12a变异体,可以有效识别5’-TATV-3’PAM序列和5’-TYCV-3’PAM序列,增加了Cas12a的靶向范围[2]。
●CRISPR/Cas12a的反式剪切
Cas12a/crRNA/DNA靶标三元复合物具有非特异性的ssDNA反式剪切活性。crRNA指导下,Cas12a识别PAM特异性识别和切割dsDNA,Cas12a发挥单链脱氧核糖核酸酶(ssDNase)活性,无差别裂解附近的ssDNA[5]。
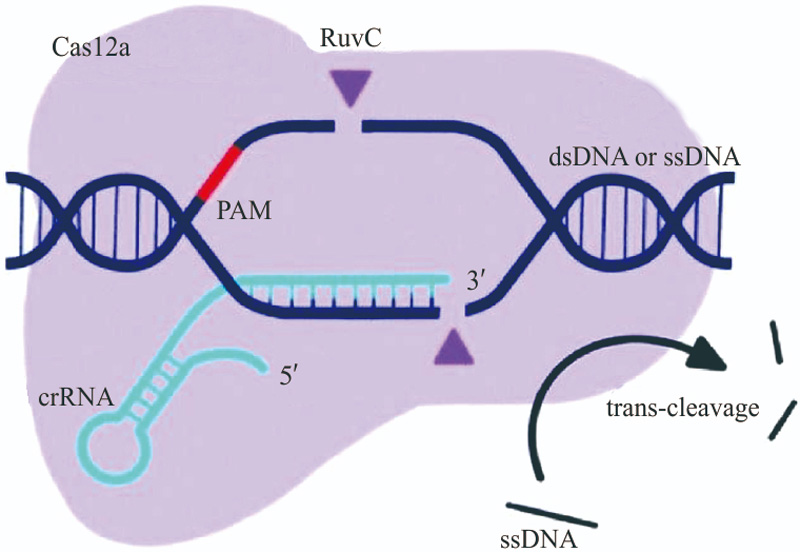
CRISPR/Cas12a系统靶向识别剪切和反式剪切示意图[6]
CRISPR/Cas12基因编辑技术在医学领域的应用
◆基于CRISPR/Cas12核酸检测方法
2018年Doudna团队将恒温扩增技术与CRISPR/Cas12a相结合开发了一种检测系统,即DETECTR,该系统可用于对样本中的微量DNA进行快速、简便的即时检测[6];同年,Cell Discovery杂志发布了HOLMES检测系统,该系统将PCR技术与CRISPR/Cas12相结合,不需要昂贵的试剂和特殊的仪器,效率高,成本低,易于进行核酸检测[7]。
先前建立的基于CRISPR/Cas12a检测方法都需要两步操作,第一步为通过恒温扩增技术或PCR技术对目的片段的扩增,第二步为基于CRISPR/Cas12的酶切反应,操作过程中需要对扩增产物进行开盖处理,这种操作不仅增加了检测所需的时间,而且操作不当会造成扩增产物泄漏污染实验室。为解决这一问题,2019年Wang等人开发了一步检测法,即Cas12avDet,并将该技术应用到了支原体的检测当中[8];上述的检测方法只能对检测结果进行定性而不能进行定量,2021年Ding等人开发了一种基于CRISPR/Cas12a的数字热启动检测方法dWS-CRISPR,将RT-DAMP和CRISPR/Cas12a相结合的一锅式检测方法,可用于临床样本中SARS-CoV-2的敏感定量检测[9]。
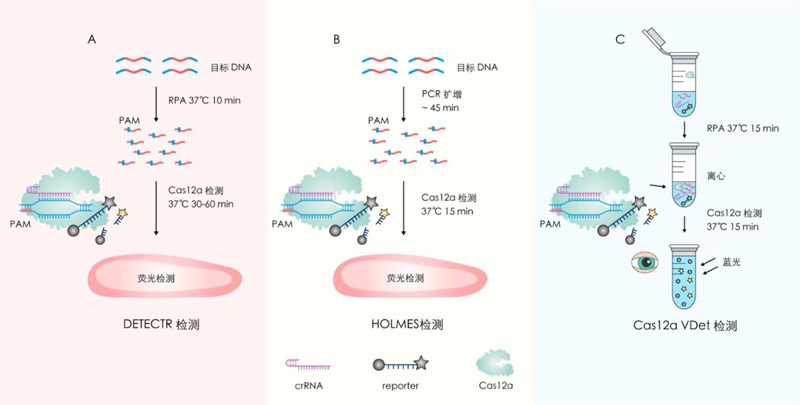
DETECTR、HOLMES、Cas12aVDet检测[10]
◆CRISPR/Cas12结合高灵敏信号转导技术的核酸检测传感器
2020年Xu等人开发了一种基于CRISPR/Cas系统的增强型电化学DNA(E-DNA)传感器[11]。2021年Liu等人开发了一种基于CRISPR/Cas12a的电化学发光生物传感器,利用L-蛋氨酸稳定金纳米团簇作为高效电化学发光(ECL)元件实现电化学发光信号转换。这种生物传感器具有较高的选择性,在不需要核酸扩增的前提下实现了对人体血液样品中HPV-16的检测,整个检测可在70min内完成[12]。同年,Liang等人将CRISPR/Cas12系统表面增强拉曼光谱(SERS)相结合建立了SERS-CRISPR(S-CRISPR)检测平台,无需核酸扩增在30-40min完成SARS-CoV-2鼻咽子标本酸检测,是一种潜在的POCT检测方法[13]。2022年Yin等人设计了一种DNA/RNA嵌合杂交探针,并将其引入到CRISPR-Cas12a/SERS系统,该技术利用DNA/RNA嵌合发夹释放的ssRNA来分解纳米团簇,不需要核酸扩增,具有高选择性和高特异性[14]。
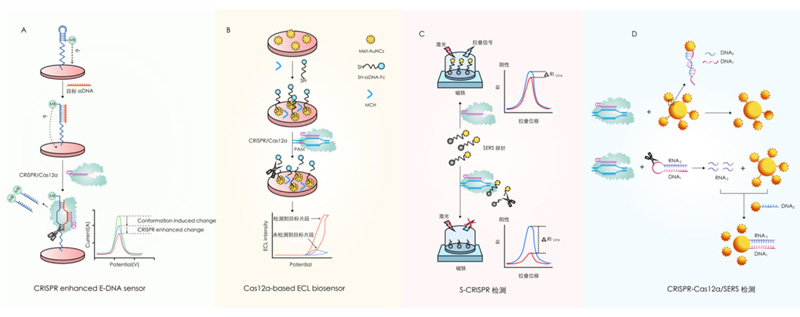
CRISPR/Cas12结合信号转导技术的核酸检测传感器[10]
◆蛋白检测
Dai等首次构建了基于CRISPR/Cas12a的蛋白检测平台,该平台将核酸适配体与靶标蛋白结合后,适配体作为Cas12a的识别底物启动其反式剪切活性,已成功用于转化生长因子β1的检测,且具有良好的灵敏度和特异性。除此之外,Zhao等开发了基于CRISPR/Cas12a的可用于检测蛋白质分析物的检测新平台,可用于临床样本中甲胎蛋白检测[6]。
CRISPR技术的快速发展为外泌体检测提供了新的思路和手段。Li等开发了基于CRISPR/Cas12a的高灵敏鼻咽癌CD109+外泌体检测平台,此外,Zhao等利用带有阻断器的适配体捕获CD63+外泌体,两者结合后适配体构象发生改变而释放阻断器,上清中的游离阻断器可激活Cas12a的反式剪切活性,进而释放荧光信号,从而实现靶标外泌体的检测[6]。Xing等使用抗体包被磁珠捕获外泌体,与适配体结合后经扩增产生可被Cas12a识别切割的长重复片段,然后非特异性切割体系中的荧光探针释放荧光信号,该平台可成功用于临床样本中外泌体表面蛋白nucleolin和程序性死亡配体1的定量检测[6]。
参考文献:
[1] Yan WX, Hunnewell P, Alfonse LE, Carte JM, Keston-Smith E, Sothiselvam S, Garrity AJ, Chong S, Makarova KS, Koonin EV, Cheng DR, Scott DA. Functionally diverse type V CRISPR-Cas systems. Science. 2019 Jan 4;363(6422):88-91.
[2] 韦涛,何滋林,纪庆超,祁艺新,常雯雯,隋宏书.CRISPR/Cas基因编辑系统研究进展[J].生命的化学,2019,39(01):39-45.
[3] 王雪亮,胡晓波.CRISPR/Cas12系统在肿瘤检测中的应用[J].临床检验杂志,2023,41(03):225-228.
[4] 董换哲, 苑宁, 张蕴哲 et al. 跨越式滚环等温扩增技术结合CRISPR/Cas12a定量检测海产品中的副溶血性弧菌[J]. 食品科学, 1-10.
[5] 孔康康. 基于CRISPR-Cas12a技术的苹果茎沟病毒快速、低成本检测体系的开发与评价[D]: 河南农业大学, 2021.
[6] 王雪亮,胡晓波.CRISPR/Cas12系统在肿瘤检测中的应用[J].临床检验杂志,2023,41(03):225-228.
[7] Li S Y, Cheng Q X, Wang J M, et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discov, 2018,4:20
[8] Wang B, Wang R, Wang D, et al. Cas12aVDet: A CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection. Anal Chem, 2019, 91(19):12156-12161.
[9] Ding X, Yin K, Li Z, et al. Sensitive quantitative detection of SARS-CoV-2 in clinical samples using digital warm-start CRISPR assay. Biosens Bioelectron, 2021, 184:113218
[10]党生,张帅,翟景波.CRISPR/Cas12a系统:核酸检测的多功能工具[J].生物化学与生物物理进展:1-13.
[11] Xu W, Jin T, Dai Y, et al Surpassing the detection limit and accuracy of the electrochemical DNA sensor through the applicatior
of CRISPR Cas systems. Biosens Bioelectron, 2020, 155:112100
[12]Liu P F, Zhao K R, Liu Z J, et al. Cas12a-based electrochemiluminescence biosensor for target amplification-free DNA detectionBiosens Bioelectron,2021, 176:112954
[13] Liang J, Teng P,Xiao W, et al. Application of the amplification-free SERS-based CRISPR/Cas12a platform in the identification ofSARS-CoV-2 from clinical samples. J Nanobiotechnology, 2021, 19(1):273
[14] Yin B, Zhang Q,Xia X, et al. A CRISPR-Cas12a integrated SERS nanoplatform with chimeric DNA/RNA hairpin guide for ultrasensitive nucleic acid detection. Theranostics,2022, 12(13):5914-5930.








