浅谈CRISPR/Cas13基因编辑技术
前言/PREFACE
CRISPR/Cas13相关研究源于2015年华人科学家张锋在Molecular Cell上发表的一篇报道[1],其后续相关报告中继续阐明了Cas13具有切割RNA的功能,正式揭开了CRISPR/Cas13系统研究和应用序幕。
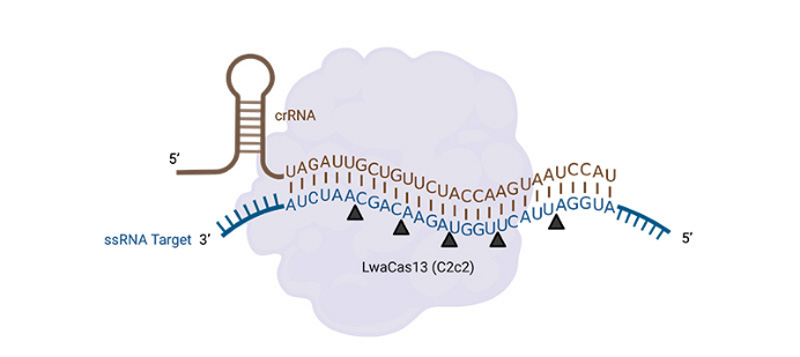
CRISPR/Cas13系统
CRISPR/Cas13系统属于CRISPR/Cas系统中的第二大类VI型,包含单一的效应蛋白质Cas13,与crispr RNA(crRNA)组装时形成一个由crRNA引导的RNA靶向效应复合物[2]。
截至目前,共发现有6种效应蛋白属于CRISPR/Cas13家族,即Cas13a、Cas13b、Cas13c、Cas13d、Cas13X和Cas13Y,Cas13X和Cas13Y是最新发现的效应蛋白,其中Cas13X是目前报道最小的RNA编辑工具。同源序列分析表明Cas13四个亚型具有低同源性,且同源序列仅限于两个高等真核生物和原核生物核苷酸(HEPN)结合域位点,HEPN可以存在于Cas13不同位置[2-4]。
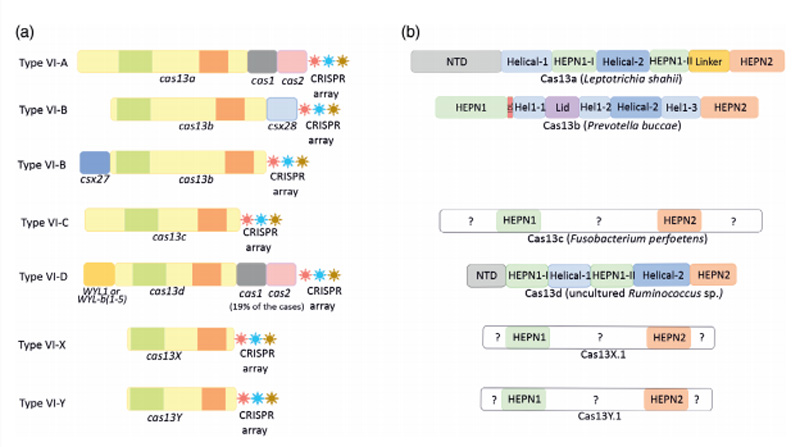
CRISPR/Cas13的分类[4]
◆CRISPR/Cas13基因编辑原理
Cas13蛋白不需要反式激活RNA(tracr RNA)或内源性核糖核酸酶(RNase),其本身具有两种不同的RNase活性,分别是将pre-crRNA预处理成成熟的crRNA和对靶标RNA进行剪切[5-7]。
Cas13由单个crRNA引导,催化自身的前体pre-crRNA生成成熟的crRNA,当crRNA-Cas13a复合物形成时,识别非靶标链中特定的前间隔序列侧翼位点(PFS,相当于Cas9识别DNA的PAM序列,位于间隔区的3’端,由A、U或C碱基组成),当Cas13-crRNA复合物识别PFS后,crRNA引导其与靶标RNA发生碱基互补配对,同时Cas13发生构象变化,使无酶切活性状态的Cas13a-crRNA复合物表现出HEPN-RNase活性,特异性地对靶标RNA进行顺式剪切,实现RNA敲除。随后还以非特异性的方式对附近的RNA进行反式剪切。根据所属亚家族的不同,Cas13切割位点不同,Cas13a切割位点是尿嘧啶或腺嘌呤;Cas13b切割位点通常是嘧啶碱基(主要是尿嘧啶);Cas13d倾向于切割尿嘧啶。CRISPR/Cas13系统的干扰活性受PFS限制,其中Cas13a效应蛋白受3′ 非G的限制,Cas13b效应蛋白受两侧PFS的限制,即5′非C和3′NAN或NNA,Cas13d效应蛋白则不受PFS限制[2,5-7]。
CRISPR/Cas13基因编辑技术在医学领域的应用
◆病毒的单分子核酸检测
依赖于Cas13非特异的RNA切割能力,Cas13已被开发为体外核酸检测的高灵敏度诊断工具[8-11]。基于Cas13a开发的SHERLOCK系统依赖于RNA荧光报告探针,其在RNA被切割之前不发射任何荧光,当Cas13a被靶标RNA激活后切割RNA荧光探针从而发出荧光,由于Cas13a强大的RNase活性,一个靶标RNA激活的Cas13a切割数以千计的RNA探针,对反应体系里的靶标RNA进行信号放大检测[8],该系统再通过等温扩增技术进一步优化,使用T7转录(RNA病毒)结合重组酶聚合酶扩增(仅扩增DNA)实现了对信号的二次放大,从而大大提高了该方法的灵敏度,对病毒核酸(DNA和RNA)实现了单分子灵敏度和单碱基特异性检测,其能够区分寨卡病毒(ZIKV)和登革热病毒(DENV),检测限低至阿摩尔级(10-18摩尔/升)[9]。2018年,Gootenberg等开发的第二代SHERLOCK,其灵敏更低(低至zeptomolar,即10-21mol水平),可实现多重靶标检测,并可通过侧流试条显色来肉眼判读结果[10]。另外,包括Cas13a-crRNA复合物在内的所有SHERLOCK组分都可以冻干,然后再水化,从而可以在现场使用SHERLOCK进行滤纸上检测[11]。Mvhrvold等开发出一种利用加热和化学还原来溶解病毒颗粒和灭活体液中高水平RNase的方法——HUDSON。HUDSON结合SHERLOCK能以极高的灵敏度(1copies/L)检测全血、血清和唾液中的病毒,并区分4种DENV血清型以及ZIKV的区域特异性流行的菌株[12]。
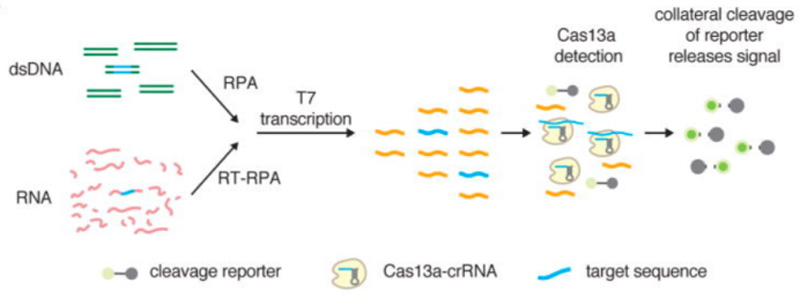
SHERLOCK工作原理[9]
◆基于RNA碱基编辑的遗传性疾病的治疗
核酸编辑有望治疗遗传疾病,特别是在RNA水平的编辑,可以敲除疾病相关序列而使致病的功能性蛋白无法翻译[13]。研究发现,来自wadei纤毛菌(Lwa)的LwaCas13a在大肠埃希菌中能够引起细胞毒性,延缓细胞生长,引发程序性细胞死亡或休眠。但是当其在人类细胞中表达时,LwaCas13a能够进行高度特异性的RNA靶向。除此之外,Cas13的HEPN结构域中的催化残基可以通过点突变失活,产生无RNA核酸酶活性的dCas13,其可以特异性结合RNA而不导致切割。dCas13b通过作用于2型RNA的腺苷脱氨酶(ADAR2)催化腺苷水解脱氨至肌苷(肌苷是在许多细胞反应中功能性模拟鸟苷的碱基),导致靶RNA上的A替换为G,该系统被命名为REPAIR,使用该系统实现了50%以上的编辑效率,还以35%和23%的效率纠正了X连锁的肾性尿崩症相关的AVPR2基因和范可尼贫血相关的FANCC基因[14]。
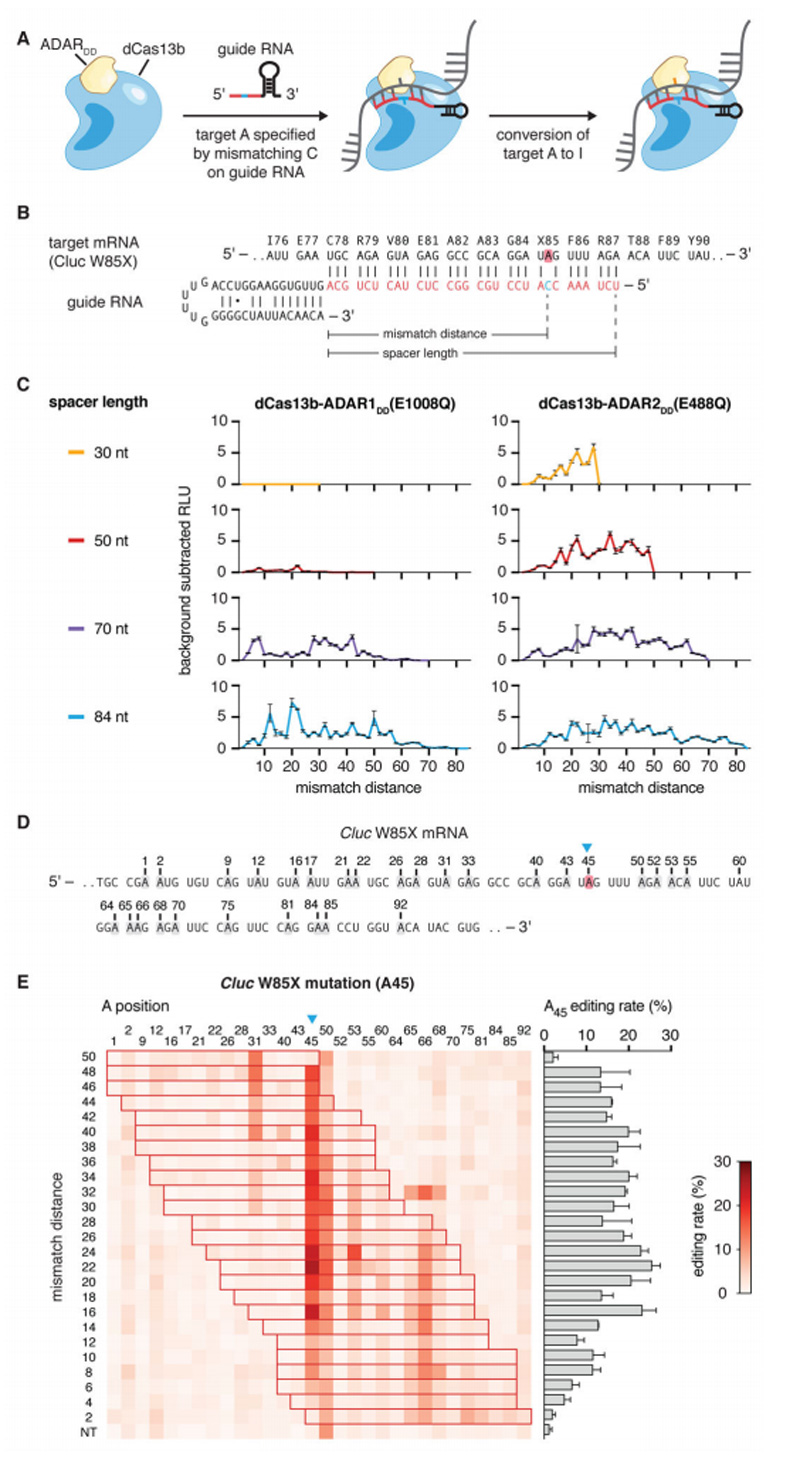
设计dCas13b-ADAR融合蛋白进行RNA编辑[14]
与DNA碱基编辑相比,RNA碱基编辑不稳定且更容易逆转,并允许对编辑过程进行精细的控制。此外DNA编辑会影响所有转录产物,而RNA编辑时编辑和非编辑的转录产物可以同时存在,这相当于实现了一定的编辑剂量控制,并避免围绕基因组编辑产生的伦理问题。DNA和RNA编辑系统也可以组合使用,当一些基因被敲除的同时,通过靶向相关基因的mRNA来敲除其他基因。同样,特定基因的转录和转录后抑制可以在单个细胞中组合,实现同时微调DNA和RNA水平的表达[8]。
◆点突变相关疾病的诊断和治疗
通过在单核苷酸多态性附近设计错配的crRNA,SHERLOCK可以通过比对不同crRNA的结果来鉴定单核苷酸多态性,对快速诊断临床相关的病毒(如ZIKV和HIV)的突变位点以及肿瘤相关的点突变具有很大的实用性。目前已有利用SHERLOCK检测方法成功鉴定出与胎儿小头畸形相关的PrM区域中的ZIKV点突变(S139N)[15]。此外,SHERLOCK可检测游离DNA(cfDNA)片段中的低频肿瘤相关的体细胞突变(EGFR L858R、BRAF V600E)[9]。
突变的KRAS是胰腺癌中已知的原癌基因,寻找可特异性地区分KRAS mRNA突变型和野生型并能有效沉默突变KRAS的靶向治疗至关重要。研究发现,CRISPR/Cas13a系统可用于胰腺癌中突变KRAS基因的靶向治疗。Cas13a蛋白和crRNA对KRAS-G12D mRNA可诱导高达70%的敲除,但对野生型KRAS mRNA几乎没有影响。Cas13a-crRNA复合物在携带KRAS-G12D突变的胰腺癌模型中能有效阻断KRAS驱动的信号通路,促进体外和体内细胞凋亡和肿瘤生长抑制[16]。
参考文献:
[1]Shmakov S,Abudayyeh O O,Makarova K S, et al. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems[J]. Molecular cell, 2015, 60(3): 385-397.
[2]陈敏洁,唐桂月,洪香娜,等.基于CRISPR-Cas13家族的RNA编辑系统及其最新进展[J].生物技术通报,2020,36(03):1-8.
[3]Xu C, Zhou Y, Xiao Q, et al. Programmable RNA editing with compact CRISPR–Cas13 systems from uncultivated microbes[J]. Nature methods, 2021, 18(5): 499-506.
[4]Kordyś M, Sen R, Warkocki Z. Applications of the versatile CRISPR-Cas13 RNA targeting system. Wiley Interdiscip Rev RNA. 2022 May;13(3):e1694.
[5]East-Seletsky A,O'Connell MR,night SC, et al.Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection[J]. Nature,2016,538(7624):270-273.
[6]Konermann S,Lotfy P,Brideau NJ,et al.Transcriptome engineering with RNA-targeting type VI-D CRISPR effectors[J].Cell.2018,173(3):665-676
[7]Zhang B,Ye Y,Ye W,et al.Two HEPN domains dictate CRISPR RNA maturation and taroet cleavage in Cas13d[J].Nat Commun.2019,10(1):2544.
[8]彭利君,许红攀,张葵.核酸检测和基因编辑的新工具:CRISPR/Cas13系统[J].临床检验杂志,2019,37(05):380-382.
[9]Gootenberg J S, Abudayyeh O O, Lee J W, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442.
[10]Gootenberg JS,Abudayyeh 00, Kellner MJ, et al.Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6[J].Science,2018,360 (6387) :439-444.
[11]East-Seletsky A, 0'connell MR, Knight SC, et al.Two distinct RNase activities of CRISPR-C2c2 enable quide-RNA processing andRNA detection[J].Nature. 2016.538 (7624) :270-273.
[12]Myhrvold C, Freje CA, Gootenberg JS, et al.Field-deployable viral diagnostics using CRISPR-Cas13[J]. Science, 2018, 360 (6387) :444-448.
[13]Abudayyeh 00, Gootenberg JS, Essletzbichler P, et al.RNA targeting with CRISPR-Cas13[J]. Nature. 2017, 550 (7675) :280-284.
[14]Cox D B T, Gootenberg J S, Abudayyeh O O, et al. RNA editing with CRISPR-Cas13[J]. Science, 2017, 358(6366): 1019-1027.
[15]Yuan L, Huang XY, Liu ZY, et al.A single mutation in the prM protein of Zika virus contributes to fetal microcephalylJj.Science, 2017,358 (6365) :933-936.
[16]Xiao Z , Liang L , Jiayan L, et al.A CRISPR-Cas13a system for eficient and specific therapeutic targeting of mutant KRAS for pancreatic cancer treatment[J].Cancer Lett, 2018,431:171-181.








