浅谈Class1 CRISPR/Cas系统
前言/PREFACE
根据CRISPR/Cas系统干扰机制的不同,分为两大类(Class1和Class2),Class1 CRISPR/Cas系统虽然研究得较少,但在自然界中含量要比Class2 CRISPR/Cas系统丰富得多,约占细菌和古细菌中CRISPR/Cas系统的90%,甚至比Class2 CRISPR/Cas系统进化得更早[1-3]。今天小编跟大家分享一下Class1 CRISPR/Cas系统。
Class1 CRISPR/Cas系统
基于不同的效应蛋白,Class1 CRISPR/Cas系统分为3个类型(Ⅰ型、Ⅲ型、Ⅳ型)、19个亚型[4-6]。
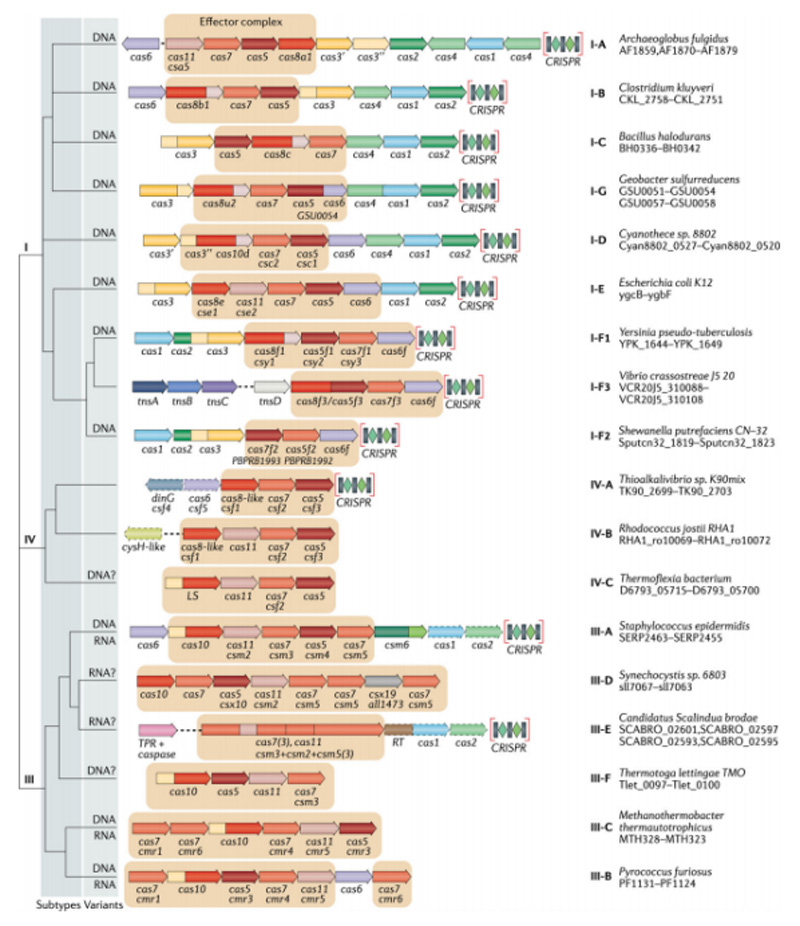
Class1 CRISPR/Cas系统分类[6]
●Ⅰ型CRISPR/Cas系统
Ⅰ型CRISPR/Cas系统是最丰富的,包含9个亚型(Ⅰ-A到Ⅰ-E、Ⅰ-F1、Ⅰ-F2、Ⅰ-F3和Ⅰ-G),其效应复合物以存在于Cas10中的RRM结构域(一种广泛存在于RNA结合蛋白中的结构域)和三个RAMP(重复相关的神秘蛋白)家族为中心。
●Ⅲ型CRISPR/Cas系统
Ⅲ型CRISPR/Cas系统包括6个亚型(Ⅲ-A-Ⅲ-F),其独特之处在它可以同时切割DNA和RNA(通过Cas7),同时靶标RNA激活Cas10发挥非特异性DNA酶的作用,产生环寡腺苷酸(cOA)第二信使。
●Ⅳ型CRISPR/Cas系统
Ⅳ型CRISPR/Cas系统是最为神秘的一类,包括3个亚型(Ⅳ-A-Ⅳ-C),其特殊之处在于,它更多存在于质粒上而非存在于染色体上。大亚基(Ⅰ型的Cas8和Ⅲ型的Cas10)的催化活性丧失和募集Cas3用于DNA切割是Ⅰ型系统进化中的关键事件,大亚基的进一步降解和适应模块(Cas1和Cas2)的丧失产生了Ⅳ型系统。
◆作用机制[3-7]
●登记病毒序列(Adaptation)
第一步:外源DNA复制频繁,会产生大量复制叉(DNA复制时在DNA链上通过解旋、解链和单链结合蛋白的结合等过程形成的Y字型结构),而当复制叉停滞时,容易发生断裂,这时细菌的修复机制就会启动,RecBCD复合物(一种在细菌中具有核心抗噬菌体功能的复合物)就结合到断裂处并向上游移动,将外源DNA解旋并打断,形成原间隔序列。
第二步:Cas1和Cas2形成的复合物会扫描体内所有的原间隔序列,如果原间隔序列末端带有原间隔序列临近基序(PAM,通常为5'-NGG-3'),复合物将会将从PAM处将原间隔序列切断,形成成熟的间隔序列。
第三步:Cas1和Cas2形成的复合物携带成熟的间隔序列识别前导序列,并将成熟的间隔序列插入前导序列与第一个间隔序列之间。因此,CRISPR序列中的间隔序列从5'到3'的排列也记录了外源遗传物质入侵的时间顺序。
第四步:最后复合物会对DNA进行修复,将打开的双链缺口闭合。这样一来,一段新的间隔序列就被添加到了基因组的CRISPR序列之中。
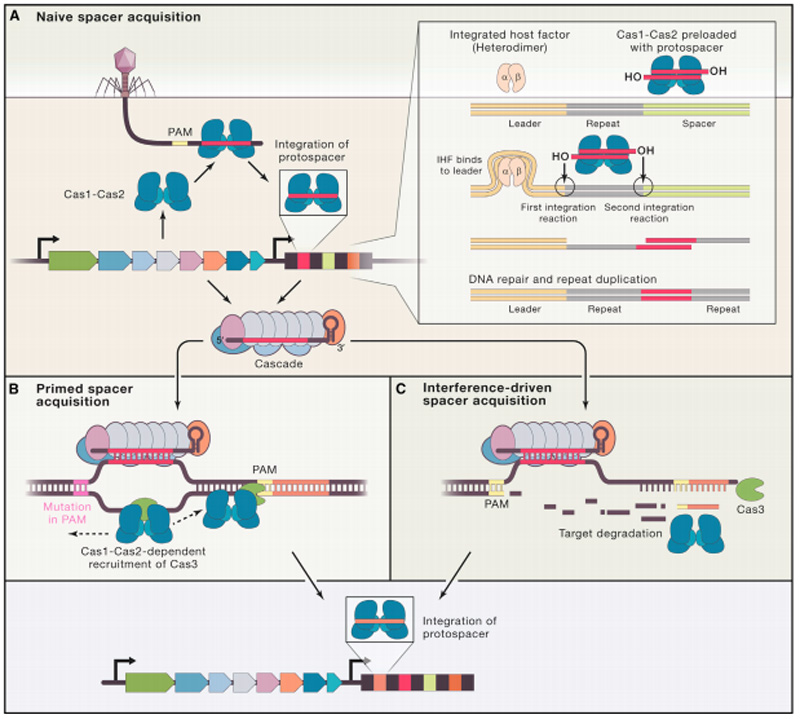
Ⅰ型CRISPR/Cas系统间隔序列的获得[7]
●CRISPR基因座的表达(Expression)
Class1 CRISPR/Cas系统基因座有两种不同表达方式,这取决于CRISPR重复序列是否含有回文序列(双链DNA中的一段倒置重复序列)。
第一步:RNA聚合酶识别前导序列中TATA-box(构成真核生物启动子的元件之一,其一致顺序为TATAATAAT),启动转录,获得包含所有间隔序列的pre-crRNA。
第二步:如果重复序列含有回文序列,pre-crRNA将会形成茎环结构,Cas6蛋白识别重复序列形成的茎环,从茎环处将pre-crRNA切断,获得带有单个间隔序列的成熟crRNA;如果重复序列不含有回文序列,Cas6蛋白将会形成二聚体,识别整个重复序列,并折叠重复序列形成类似茎环的凸起,从凸起处将pre-crRNA切开,获得成熟的crRNA。
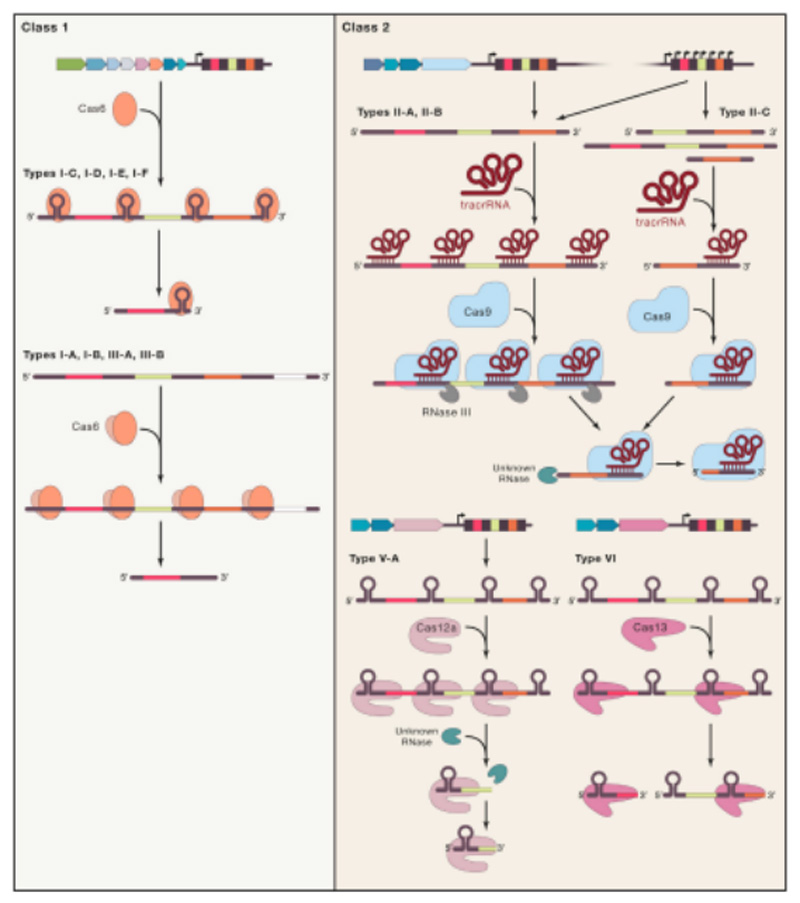
Class1和Class2 CRISPR/cas系统的crRNA成熟途径[7]
●CRISPR-Cas系统靶向干扰(Interference)
Class1 CRISPR/Cas系统靶向干扰有两种不同方式。
■ Ⅰ型CRISPR/Cas系统靶向干扰
第一步:Cas6蛋白识别crRNA茎环,并招募一系列Cas蛋白形成复合物。有时Cas6蛋白在加工pre-crRNA形成crRNA后并不会脱落,而直接进行靶向干扰。
第二步:复合物携带crRNA扫描体内所有DNA序列,并识别PAM,当携带crRNA的复合物识别到PAM时,复合物将会停止移动并将PAM下游DNA双链解旋,此时crRNA会尝试与上游DNA序列配对,当crRNA与目标链匹配时,复合物将会诱导上游DNA序列形成R环。
第三步:Cas3核酸酶的将会识别DNA序列形成的R环并切断识别位置的互补链。
■ Ⅲ型CRISPR/Cas系统靶向干扰
第一步:Cas5蛋白识别crRNA残留的5'端重复序列残基,并招募一系列Cas蛋白形成复合物。
第二步:复合物携带crRNA扫描体内所有的RNA序列,crRNA会尝试与转录的RNA序列配对,当crRNA与目标链匹配时,复合物中Cas10亚基将会结合到正在转录的RNA上游并将正在转录的DNA双链切断,Cas10将激活活性可以将ATP转化为环状寡腺苷酸(cOA)。
第三步:复合物中的Cas7亚基将会裂解与crRNA匹配的转录片段。cOA作为第二信使激活Csm6核糖核酸酶,Csm6核糖核酸酶将非特异性的降解RNA。
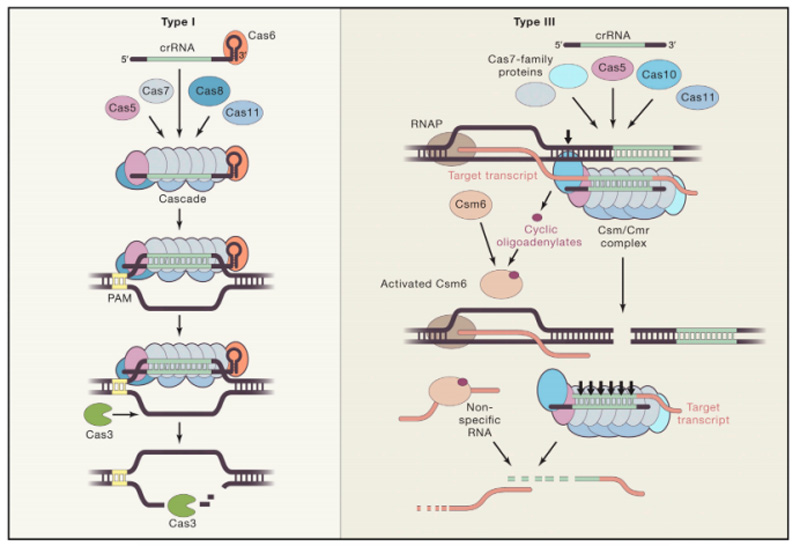
Class1 CRISPR/Cas系统的干扰途径[7]
Class1 CRISPR/Cas系统应用
关于Class1 CRISPR/Cas系统的应用研究相对较少,主要集中于Ⅰ型和Ⅲ型CRISPR/Cas系统,已开发出CONAN(Cas3-operated nucleic acid detection)检测技术和MORIARTY检测技术[8]。
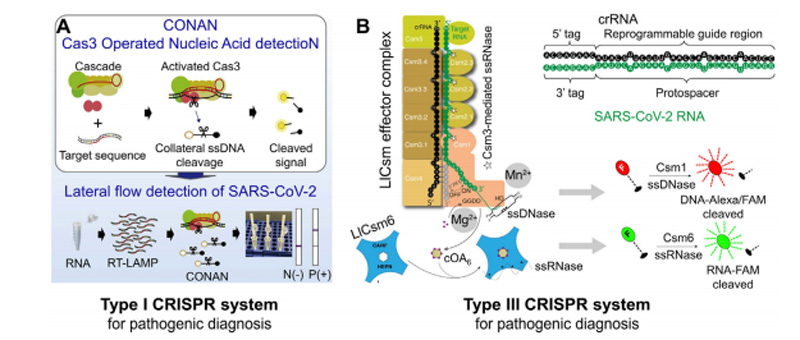
基于Class1 CRISPR/Cas的病原体核酸诊断[8]
◆Ⅰ型CRISPR/Cas系统
有研究描述Ⅰ-E型CRISPR-Cas3也具有反式切割活性,主要利用Cascade效应模块(由Cas5、Cas6、Cas7、Cas8和Cas11等多种蛋白组成)、Cas3蛋白和crRNA组成的复合物,在与靶标结合后,完成单链DNA探针的切割。而且有研究表明,Cas3-Cascade/crRNA 效应复合物必须与靶标DNA形成完整的R环结构,才能利用Cas3蛋白完成切割,这一特性可以防止Cas3过早与DNA结合,引起非特异性切割。基于此新开发的CONAN检测技术对单碱基区分具有很高的特异性,不仅成功检测出临床样本中SARS-CoV-2,还可以特异性检测甲型流感病毒变体中的单碱基对突变[9]。此外,Dolan等人首次成功使用Ⅰ型CRISPR/Cas系统CRISPR/Cas3在人胚胎干细胞(hESCs)和HAP1细胞中实现基因组大片段删除(如成功敲除了长达100Kbp的DNA超长片段),在hESCs和HAP1细胞中的编辑效率分别达到13%和30-60%。该研究表明Ⅰ型CRISPR/Cas系统在真核系统中具有巨大的基因编辑潜力和应用价值。CRISPR/Cas3与传统编辑系统相相比较,Cas3不是仅仅将DNA双链进行切割,还会连续消除DNA片段,最多可长达100Kbp碱基对。相比于CRISPR/Cas9,CRISPR/Cas3可以更加便捷及更高效率实现对基因组大片段的敲除。因此,利用该基因编辑系统可对人类基因组非编码遗传元件进行大片段的基因敲除,从而探索基因组片段的作用。在未来该技术可以用来消除寄生或疾病的遗传因素,或者引入远距离的表观遗传修饰。这种基于Ⅰ型CRISPR/Cas系统在真核基因编辑中的成功应用,极大地扩展了基因组编辑工程工具包[10]。
◆Ⅲ型CRISPR/Cas系统
Ⅲ型CRISPR/Cas系统在极端嗜热微生物的基因编辑中展现出了超越Cas9系统的独特优势,2016年Li等在极端嗜热古菌S. islandicus中率先开发了基于Ⅲ型CRISPR/Cas系统的基因编辑技术,该技术通过在质粒上构建表达特异crRNA的mini-CRISPR实现精确的基因编辑。与基因编辑方法类似,Zebec等将Ⅲ型CRISPR/Cas系统用于基因沉默,但是需要在设计时确保靶标序列的3’端与成熟crRNA的5’handle序列完全匹配[11]。
近期,Raymond等人CRISPR/Cas系统基于Ⅲ型CRISPR/Cas系统开发了一种新的诊断工具SCOPE(Screening using CRISPR Oligoadenylate- Perceptive Effectors),用于从人鼻拭子样本中特异性检测SARS-CoV-2[12]。
除此之外,基于Ⅲ-A型系统的方法开发的MORIARTY检测技术也可用于检测SARS-CoV-2,在30min内达到2000拷贝/μL(免扩增)和60拷贝/μL(等温扩增)的检测限,并且灵敏度高,反应条件灵活。与实时荧光定量PCR方法相比,MORIARTY无需热循环,且易与已经开发的POCT技术结合[8,13]。
◆Ⅳ型CRISPR/Cas系统
Ⅳ型CRISPR/Cas系统对于同一宿主的其他质粒具有极强的靶向性,因此有可能会介导宿主中不同质粒之间的竞争。有研究表明该系统可以靶向其他质粒中的转移基因,这暗示着Ⅳ型CRISPR/Cas系统可能具备对抗携带耐药基因质粒的能力[14]。
参考文献:
[1] Brouns, S. J. J., Charpentier, E., Cheng, D., et al (2020) Evolutionary classification of CRISPR–Cas systems: a burst of Class 2 and derived variants. Nat. Rev. Microbiol. 18, 67–83.
[2] Makarova, K. S., Wolf, Y. I., Alkhnbashi, O. S., et al(2015) An updated evolutionary classification of CRISPR–Cas systems.Nat. Rev. Microbiol. 13, 722–736.
[3] Burstein, D., Sun, C. L., Brown, C. T., et al. (2016) Major bacterial lineages are essentially devoid of CRISPR-Cas viral defence systems. Nat.Commun. 7, 10613.
[4] Liu TY, Doudna JA. Chemistry of Class 1 CRISPR-Cas effectors: Binding, editing, and regulation. J Biol Chem. 2020 Oct 16;295(42):14473-14487.
[5] Makarova KS, Zhang F, Koonin EV. SnapShot: Class 1 CRISPR-Cas Systems. Cell. 2017 Feb 23;168(5):946-946.e1.
[6]Makarova KS, Wolf YI, Iranzo J, et al. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol. 2020 Feb;18(2):67-83.
[7] Hille F, Richter H, Wong SP, et al, Charpentier E. The Biology of CRISPR-Cas: Backward and Forward. Cell. 2018 Mar 8;172(6):1239-1259.
[8] Yang H, Zhang Y, Teng X, Hou H, Deng R, Li J. CRISPR-based nucleic acid diagnostics for pathogens. Trends Analyt Chem. 2023 Mar;160:116980.
[9] 杜瑶,高宏丹,刘家坤等.CRISPR-Cas系统在病原核酸检测中的研究进展[J/OL].合成生物学:1-15[2023-10-18].
[10] Dolan AE, Hou Z, Xiao Y, Gramelspacher MJ, Heo J, Howden SE, Freddolino PL, Ke A, Zhang Y. Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas. Mol Cell. 2019 Jun 6;74(5):936-950.e5.
[11] Liu T, Pan S, Li Y, Peng N, She Q. Type III CRISPR-Cas System: Introduction And Its Application for Genetic Manipulations. Curr Issues Mol Biol. 2018;26:1-14.
[12] Steens JA, Zhu Y, Taylor DW, et al. SCOPE enables type III CRISPR-Cas diagnostics using flexible targeting and stringent CARF ribonuclease activation. Nat Commun. 2021 Aug 19;12(1):5033.
[13] Sridhara S, Goswami HN, Whyms C, Dennis JH, Li H. Virus detection via programmable Type III-A CRISPR-Cas systems. Nat Commun. 2021 Sep 27;12(1):5653.
[14] Pinilla-Redondo R, Mayo-Muñoz D, Russel J, et al. Type IV CRISPR-Cas systems are highly diverse and involved in competition between plasmids. Nucleic Acids Res. 2020 Feb 28;48(4):2000-2012.








